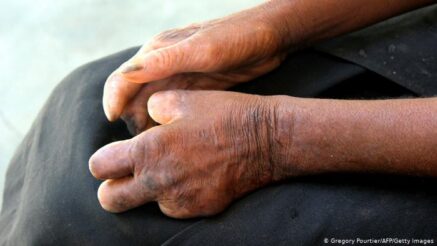
Los biomarcadores sanguíneos pueden ser útiles en estrategias para controlar la lepra, una infección crónica desatendida que afecta a miles de personas en países endémicos. La detección temprana de la lepra es la piedra angular para brindar un tratamiento oportuno que interrumpa la transmisión de Mycobacterium leprae (M. leprae) y prevenga discapacidades físicas y deformidades. Se calcula que los años de vida perdidos por discapacidad a causa de la lepra oscilan entre 2 y 6 de cada 10.000 personas, siendo más alto en hombres que en mujeres.
La aparición y la percepción de los signos y síntomas de la lepra pueden tardar varios años mientras M. leprae se replica en general, especialmente en personas que carecen de una respuesta inmune celular adecuada, que es esencial para suprimir esta bacteria intracelular obligada. Por lo tanto, la cadena de transmisión persiste sin previo aviso. Por el contrario, las personas con una respuesta inmune adecuada, muestran una carga bacteriana imperceptible y pueden tener pocas lesiones inusuales, lo que dificulta el diagnóstico con las pruebas serológicas y moleculares disponibles actualmente.

Al final, en estos casos prevalece el diagnóstico clínico y epidemiológico, apoyándose en la experiencia de los médicos. Por lo tanto, la lepra es una enfermedad compleja con un amplio espectro de características clínicas e inmunológicas para las que existe una necesidad urgente de pruebas confiables para la pronta identificación de casos en países endémicos.
En este contexto, informe sobre un biomarcador sanguíneo, la firma RISK4LEP que permitió identificar a las personas afectadas por la lepra hasta 5 años después de la inscripción entre una gran cohorte de contactos domésticos (HC) de personas afectadas por la lepra. Este es un estudio de desarrollo traslacional integral que integró transcriptómica y genómica, con parámetros epidemiológicos y clínicos. Los autores llevan al lector a través de todo el proceso de desarrollo hacia la firma final de 4 genes (MT-ND2, REX1BD, TPGS1 y UBC), designada como RISK4LEP, que tiene una sensibilidad y especificidad aceptables para predecir el desarrollo de lepra límite en individuos en alto riesgo de lepra en Bangladesh. El modelo incluyó validación interna utilizando el conjunto de genes seleccionados de aquellos con sondas disponibles comercialmente para PCR cuantitativa de transcripción inversa.
El estudio sistemático de los perfiles de expresión génica obtenidos a partir de la transcripción de material genético en la célula, mediante la secuenciación del ácido ribonucleico (ARN) y el análisis bioinformático está potenciando el desarrollo de nuevos biomarcadores para muchas enfermedades. La investigación de los cambios en los patrones de expresión en respuesta a los patógenos, tanto a nivel del transcriptoma como del proteoma, nos ha ayudado a obtener una comprensión más profunda de la relación huésped-patógeno y los procesos de la enfermedad. Sabemos que la replicación y la progresión de la enfermedad dependen de la transcripción celular del huésped y la regulación génica en las células diana específicas de M. leprae. Tanto los factores bacterianos como los del huésped están implicados en esta regulación diferencial.
Las matrices de genes y los análisis de transcriptomas podrían arrojar luz sobre por qué algunas personas infectadas permanecen asintomáticas. Un modelo similar al presentado por Tió-Coma et al. podría usarse para buscar biomarcadores sanguíneos para predecir los eventos inmunoinflamatorios conocidos como reacciones leprosas o progresión del daño nervioso. Estas dos complicaciones reducen la calidad de vida del paciente y pueden provocar discapacidades, por lo que los pacientes en riesgo se beneficiarían de una intervención preventiva.
En el marco del plan operativo de la Estrategia mundial contra la lepra , HC y / o contactos sociales deben ser examinados para detectar lepra poco después de la detección del caso índice, y luego, se supone que deben ser examinados anualmente durante 5 años. Sin embargo, la vigilancia de contactos durante un período tan largo es un desafío para el servicio primario de salud y costosa para el sistema de salud, especialmente en áreas de bajos recursos. Además, aunque se espera que las personas expuestas infectadas tengan un mayor riesgo de contraer lepra, es posible que no evolucionen para tener la enfermedad. Por lo tanto, existe un gran potencial para encontrar una firma genética predictiva de la lepra en la HC. Puede permitir identificar a las personas que podrían beneficiarse del uso de medidas preventivas y ayudaría a orientar una intervención más específica.
Una tarea importante es garantizar que los biomarcadores prometedores utilicen tecnología y métodos que otros investigadores puedan reproducir fácilmente y en diferentes entornos. Lo ideal es que los ensayos utilicen plataformas que se puedan transferir o adaptar fácilmente para su uso en la clínica y el campo. La incorporación de firmas genéticas en la toma de decisiones clínicas es un proceso lento y limitado en varios aspectos. Los estudios de validación independientes son fundamentales para evaluar aún más la precisión predictiva y la utilidad de la firma RISK4LEP en la práctica clínica y proporcionar la evidencia de alta calidad necesaria de su utilidad predictiva. Es importante notar que todos los casos incidentes desarrollaron lepra indeterminada o formas del espectro tuberculoide de la enfermedad. Aunque este tipo de lepra se diagnostica con mayor frecuencia en cohortes de HC , es de mayor interés predecir el desarrollo de las formas lepromatosas más graves y transmisibles de la enfermedad. Por lo tanto, se necesitan estudios prospectivos para verificar tanto la validez clínica como la utilidad en diferentes entornos y muestras de población para corroborar la importancia predictiva. Esperamos que la prueba pueda lograr un nivel suficiente de rendimiento de la muestra y asequibilidad del ensayo para permitir su transferencia al punto de atención e incorporación para su uso en condiciones programáticas.
Fuente: https://www.thelancet.com/journals/ebiom/article/PIIS2352-3964(21)00206-1/fulltext